Por Douglas Jordan con el aporte del Dr. Terrence Tumpey y Barbara Jester
- Antecedentes: El legado mortal de la pandemia de 1918 y su importancia para los esfuerzos mundiales para prepararse ante las amenazas de futuras pandemias.
- Parte 1 – Descubrir al asesino perdido: la historia de una eterna búsqueda del cazador de virus para descubrir el virus de influenza pandémica más mortal en la historia humana.
- Parte 2 – Armar el proyecto : la historia de cómo un equipo de científicos estadounidenses decodificó y armó el genoma del virus de 1918.
- Parte 3 – La reconstrucción: la historia de cómo un microbiólogo de los CDC reconstruyó el virus vivo de la influenza pandémica de 1918 en un laboratorio seguro de los CDC para descifrar sus secretos y proteger contra las futuras pandemias.
- Parte 4 – Lecciones del pasado: cómo avanzó el mundo desde la pandemia de 1918 y los retos que suponen las futuras pandemias.
- Referencias
Antecedentes
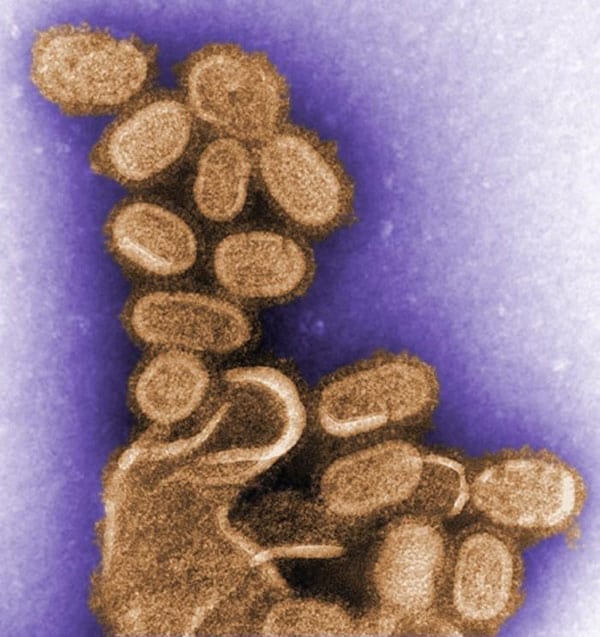
Una imagen a color del virus de 1918 tomada a través de un microscopio electrónico de transmisión (TEM, por sus siglas en inglés). El virus de 1918, que causó la pandemia de influenza más mortal registrada en la historia humana, se cobró la vida de alrededor de 50 millones de personas en todo el mundo. Foto por: C. Goldsmith – Biblioteca de imágenes de salud pública n.° 11098.
El 100.° aniversario de la pandemia de 1918 y el 10.° aniversario de la pandemia de influenza H1N1 2009 son hitos históricos que ofrecen una oportunidad para reflexionar sobre un trabajo revolucionario que dio lugar al descubrimiento, la secuenciación y la reconstrucción del virus de la influenza pandémica de 1918. Este esfuerzo conjunto potenció el entendimiento de la pandemia de influenza más mortal en la historia moderna y ha permitido que la comunidad de salud pública a nivel mundial se prepare para las pandemias contemporáneas, como la pandemia de influenza H1N1 2009, y las amenazas de futuras pandemias.
La pandemia de la influenza H1N1 1918, muchas veces llamada «Influenza española», se cobró la vida 50 millones de personas en todo el mundo, que incluye alrededor de 675 000 personas en los Estados Unidos.1,2,3,4 Una característica atípica de este virus fue la alta tasa de muertes que causó entre los adultos sanos de 15 a 34 años de edad.3 La pandemia disminuyó la expectativa de vida en los Estados Unidos por más de 12 años.3 No se ha observado una tasa de mortalidad equiparable durante cualquiera de las temporadas de influenza que se conocen o pandemias que ocurrieron antes o después de la pandemia de 1918.3
La gravedad de este virus único desconcertó a los investigadores durante décadas y generó preguntas tales como: «¿Por qué fue tan mortal el virus de 1918?», «¿Dónde se originó el virus?» y «¿Cuál es el aprendizaje de la comunidad de salud pública del virus de 1918 para prepararse mejor y defenderse de futuras pandemias?» Todas estas preguntas dieron lugar a un grupo de expertos investigadores y cazadores de virus para buscar el virus perdido de 1918, realizar la secuenciación de sus genomas, recrear el virus en un entorno de laboratorio regulado y altamente seguro de los CDC y por último estudiar sus secretos para prepararse mejor ante futuras pandemias. Lo siguiente es un recuento histórico de estos esfuerzos, con referencias y descripciones de los aportes realizados por todos los destacados hombres y mujeres involucrados.
Descubrir al asesino perdido

Sitio de la fosa común en Brevig Mission, Alaska, donde fueron enterrados 72 de los 80 habitantes de un pequeño pueblo que sucumbieron al virus mortal de la pandemia de 1918. Foto por: Angie Busch Alston.

Esta foto de 1951 muestra a Johan Hultin (a la izquierda) y a sus colegas universitarios durante su primer intento por obtener el virus de 1918 de los cuerpos de las víctimas sepultadas en el cementerio de Brevig Mission. Foto por: Johan Hultin
Durante décadas, el virus de 1918 ha quedado en el olvido como una reliquia de una época cuando los conocimientos acerca de los patógenos infecciosos y las herramientas para estudiarlos aún estaban en sus etapas incipientes. Después de la pandemia de 1918, generaciones de científicos y expertos en salud pública se quedaron únicamente con evidencias epidemiológicas del poder letal del virus de la pandemia de 1918 y el impacto nocivo que tuvo en la población mundial. Un pequeño pueblo costero de Alaska llamado Brevig Mission se convertiría en un testimonio de este legado mortal y el lugar crucial para el posible descubrimiento del virus de 1918.
Actualmente, menos de 400 personas viven en Brevig Mission, pero en el otoño de 1918, cerca de 80 adultos vivían allí, principalmente los Inuits. Si bien existen diferentes relatos sobre cómo llegó el virus de 1918 a este pequeño pueblo, ya sea por comerciantes de una ciudad aledaña que se trasladaban en trineos tirados por perros o por un personal local encargado de la correspondencia, está documentado el impacto que causó en la población de este lugar. Durante un lapso de cinco días entre el 15 y el 20 de noviembre de 1918, la pandemia de 1918 se cobró la vida de 72 de los 80 habitantes adultos del pueblo.
Posteriormente, por orden del gobierno local, se creó una fosa común señalizada solo por pequeñas cruces blancas en una colina cerca del pueblo, un monumento sombrío para una comunidad completamente arrasada. La fosa quedó completamente congelada en permafrost y se mantuvo intacta hasta 1951. Ese año, Johan Hultin, un microbiólogo sueco de 25 años y estudiante de posgrado de la Universidad de Iowa, se aventuró en una expedición a Brevig Mission con la esperanza de encontrar el virus de 1918 y en el proceso poder descubrir nuevos datos y responder preguntas. Hultin creía que dentro de ese cementerio preservado encontraría vestigios del virus de 1918 congelados en los tejidos de los pobladores que murieron a causa de este virus.
En 1951, Hultin obtuvo el permiso de los ancianos del pueblo para excavar el cementerio de Brevig Mission. Con la ayuda de varios de sus colegas universitarios, Hultin instaló un sitio de excavación sobre la fosa. La excavación llevó días ya que Hultin tuvo que hacer hogueras para descongelar la tierra y poder excavar. Al cabo de dos días, Hultin se topó con el cuerpo de una pequeña niña —su cuerpo aún estaba intacto, llevaba puesto un vestido azul y lazos rojos que adornaban su cabello5. Finalmente, Hultin obtuvo tejido pulmonar de otros cuatro cuerpos enterrados en el lugar, pero las limitaciones logísticas y tecnológicas de aquella época terminarían siendo imbatibles.
En una conversación que Hultin tuvo décadas después con el microbiólogo de los CDC, el Dr. Terrence Tumpey, (ver parte III: la reconstrucción), Hultin explicaría cómo durante el viaje de vuelta desde Alaska hasta la Universidad de Iowa en un avión de propulsión DC-3 se vieron obligados a tener que parar varias veces para repostar combustible. En cada parada, Hultin, siempre ingenioso, bajaba del avión e intentaba volver a congelar las muestras de pulmón utilizando el dióxido de carbono del matafuego.
El ruido que hacía con esto aparentemente atraía las miradas desconcertadas de los pasajeros que lo acompañaban y de los curiosos. Una vez de regreso en Iowa, Hultin intentó inyectar el tejido pulmonar en huevos de gallina con la intención de que el virus se reproduzca5. Pero no fue así. Al final, probablemente como era de esperarse, Hultin no pudo recuperar el virus de 1918 en su primer intento.

Una foto de Johan Hultin trabajando en el laboratorio en 1951. El primer intento de Hultin por recuperar el virus de 1918 fracasó. Nota: Actualmente utilizar la boca para colocar el virus dentro de una pipeta no está considerada una práctica segura de laboratorio. Las prácticas seguras de laboratorio han avanzado muchísimo en los tiempos modernos. Foto por: Johan Hultin.

Una foto del Dr. Jeffery Taubenberger y la Dra. Ann Reid analizando una secuencia genética del virus de 1918. A ellos se les atribuye la secuenciación del genoma del virus de 1918. Foto por: exposición en línea del Museo Nacional de Salud y Medicina – MIS 377212.
Esto no ocurriría hasta 46 años después, en 1997, cuando Hultin tendría otra oportunidad para buscar el virus de 1918. Ese año, Hultin se encontró con un artículo de la publicación Science escrito por Jeffery Taubenberger et al. titulado “Initial Genetic Characterization of the 1918 “Spanish” Influenza Virus”.6 En ese momento, el Dr. Taubenberger era un joven patólogo molecular que trabajaba en el Armed Forces Institute of Pathology en Washington, D.C.
En el artículo, Taubenberger y su equipo describían su trabajo inicial para realizar la secuenciación de una parte del genoma del virus de 1918. El genoma es la lista completa de las instrucciones genéticas que conforman un organismo, similar a un proyecto utilizado para construcción. Muchas personas están familiarizadas con el concepto de ADN, el cual consiste en una cadena doble y determina las características genéticas fundamentales de prácticamente todos los organismos vivos. Sin embargo, el genoma de un virus de influenza consiste en una sola cadena de ARN. El equipo de Taubenberger había extraído el ARN del virus de 1918 del tejido pulmonar obtenido de un hombre de 21 años de edad, miembro del servicio militar de EE. UU. apostado en Fort Jackson, Carolina del Sur. El soldado fue internado en el hospital del campamento el 20 de septiembre de 1918, con un diagnóstico de infección por el virus de influenza y neumonía. Falleció al cabo de seis días el 26 de septiembre de 1918 y se le tomó una muestra de tejido pulmonar que se preservó para estudios posteriores.
De este tejido, el grupo de Taubenberger fue capaz de realizar la secuenciación de nueve fragmentos de ARN viral de cuatro de los ocho segmentos genéticos del virus. Este trabajo no representó una secuencia completa de todo el genoma del virus de 1918, pero proporcionó una imagen más clara del virus pandémico como nunca antes. De acuerdo con los datos de la secuencia del virus de 1918 que Taubenberger reunió en 1997, él y sus compañeros de investigación en un principio afirmaron que el virus de 1918 fue un nuevo virus de influenza A (H1N1) que perteneció a un subgrupo de virus provenientes de cerdos y humanos, en contraposición a las aves.6 No obstante, aún quedaba mucho por aprender acerca del virus.
Después de leer el artículo de Taubenberger, Hultin nuevamente se inspiró e intentó recuperar el virus de 1918. Hultin le escribió una carta a Taubenberger. En esta le preguntaba si le interesaría que él regresara a Brevig Mission y obtuviera nuevamente tejidos pulmonares de las víctimas del virus de 1918 que fueron enterradas bajo el suelo de permafrost en Alaska. Taubenberger le devolvió una llamada y aceptó su propuesta. Una semana después, Hultin partió otra vez hacia Brevig Mission con escasas herramientas para realizar el trabajo. Le pidió prestadas a su esposa las tijeras de jardín para realizar la excavación.
Habían pasado cuarenta y seis años desde el primer viaje de Hultin al cementerio y ahora tenía 72 años de edad. Otra vez solicitó un permiso al ayuntamiento del pueblo para excavar las tumbas, el cual se lo concedieron, y contrató a lugareños para que lo ayuden con el trabajo. Los gastos de este viaje corrieron por cuenta de Hultin y le costó cerca de $3,200.7 La excavación duró aproximadamente cinco días, pero esta vez Hultin logró un hallazgo extraordinario.
Encontraron el cuerpo de una mujer Inuit enterrado y conservado por el permafrost a casi 7 pies de profundidad a la que Hultin llamó “Lucy”. Hultin descubriría que Lucy había sido una mujer obesa que probablemente había muerto a los 20 y tantos a causa de las complicaciones por el virus de 1918. Los pulmones estaban perfectamente congelados y conservados por el permafrost de Alaska. Hultin se los extirpó, los colocó dentro de un líquido conservante y luego se los envió por separado a Taubenberger y sus colegas investigadores, incluida la Dra. Ann Reid, del Armed Forces Institute of Pathology.5 Diez días más tarde, Hultin recibió una llamada de los científicos para confirmar —quizás para sorpresa de todos— que material genético del virus de 1918 con resultado positivo había sido obtenido del tejido pulmonar de Lucy.

Una foto de Johan Hultin en el cementerio de Brevig Mission en 1997, 46 años después de su primer intento para rescatar el virus de la influenza pandémica de 1918. Hultin notó que faltaban las pequeñas cruces que anteriormente cubrían el lugar, entonces construyó dos cruces enormes (como se muestra arriba) en el taller de carpintería de una escuela local para marcar el área del cementerio. Foto por: Johan Hultin.

A los 72 años de edad, Johan Hultin en el lugar donde se encuentran las tumbas durante su segundo viaje a Brevig Mission en 1997. Foto por: Johan Hultin.

Una foto de Johan Hultin excavando un cuerpo en las tumbas de Brevig Mission. En el centro de la imagen, pueden verse las tijeras de jardín que Hultin le pidió prestadas a su esposa para realizar la excavación. Foto por: Johan Hultin.
Armar el proyecto
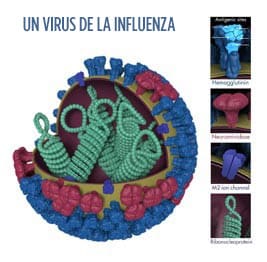
Esta es la imagen de un virus de la influenza. La hemaglutinina (HA) es una proteína superficial del virus que permite que ingrese un virus de la influenza e infecte una célula sana. Foto por: Dan Higgins, CDC.
El impacto inicial de este descubrimiento sería descrito por primera vez en un documento en febrero de 1999 titulado “Origin and evolution of the 1918 “Spanish” influenza virus hemagglutinin gene,” de Ann Reid et al., publicado en la revista Proceedings of the National Academy of Science (PNAS). 8 Hultin fue reconocido como coautor. En este documento, los autores describieron sus esfuerzos para realizar la secuenciación (es decir, la caracterización) del gen de la hemaglutinina “HA” del virus de 1918.
El gen de HA de un virus de influenza determina las propiedades de las proteínas superficiales de la HA del virus. Estas proteínas superficiales de la HA permiten que ingrese un virus de la influenza e infecte las células de las vías respiratorias sanas. La HA además es atacada por los anticuerpos producidos por el sistema inmunitario para combatir la infección. Las vacunas contra la influenza modernas inhiben la función de la HA exclusiva del virus de la influenza (un hecho que el Dr. Peter Palese, virólogo, mencionó posteriormente en este artículo, que ayudó a llevar a cabo).
En el estudio de 1999, los autores lograron realizar la secuenciación de toda la secuencia de genes de la HA del virus de 1918. Para concretar esto, los autores utilizaron fragmentos de ARN del virus obtenido del cuerpo del hombre de 21 años, miembro del servicio militar en Fort Jackson, descrito anteriormente, del cuerpo de «Lucy» de Brevik Mission, y de una tercera persona, un hombre de 30 años de edad, miembro del servicio militar apostado en Camp Upton, Nueva York. Este hombre fue ingresado al hospital militar con un cuadro de influenza el 23 de septiembre de 1918, la enfermedad avanzó rápidamente y falleció el 26 de septiembre de 2018 por una insuficiencia respiratoria aguda.
Los resultados de la secuenciación sugirieron que el predecesor del virus de 1918 infectó a seres humanos entre 1900 y 1915. Los doctores Reid y Taubenberger notaron que el gen de la HA de 1918 tenía una cantidad de adaptaciones a mamíferos a diferencia de adaptaciones a aves y se parecía más al de los humanos o los cerdos según el método de análisis. El análisis filogenético, el cual es utilizado para agrupar los virus de la influenza conforme con su desarrollo revolucionario y diversidad, ubicó a la HA del virus de 1918 dentro de la raíz del clado mamífero. Esto significa que probablemente fue un predecesor o estaba estrechamente relacionado con los virus de la influenza anteriores que eran conocidos por infectar a los mamíferos. Sin embargo, los autores consideraron que el virus probablemente obtenía su HA de los virus de la influenza aviar, pero no estaban seguros de cuánto tiempo los virus se estarían adaptando en un organismo hospedador mamífero antes de surgir en forma de pandemia.
Según los autores, la cepa existente con la cual las secuencias del virus de 1918 estaban más estrechamente relacionadas fue la «A/sw/Iowa/30», la cepa de la influenza porcina clásica más antigua. Los autores observaron que las cepas del virus de la influenza aviar contemporáneo difieren considerablemente del virus de la pandemia de 1918 y lamentablemente las cepas de la influenza aviar más antiguas de la época de la pandemia de 1918 no estaban disponibles para ser estudiadas. Los autores también notaron que el HA1 del virus de 1918 tenía solo cuatro sitios de glicosilación, lo cual difiere de la HA humana moderna que ha acumulado hasta cinco sitios de glicosilación a través del proceso de variación antigénica. La variación antigénica del virus se refiere a pequeños cambios en los genes de los virus de influenza que ocurren constantemente con el tiempo a medida que el virus se reproduce. La variación antigénica del virus es el motivo por el cual existe una temporada de influenza cada año y además es el motivo por el cual las personas pueden contraer la influenza varias veces a lo largo de sus vidas.
Se cree que los sitios de glicosilación son necesarios para la función de los virus de influenza y se considera que la incorporación de otros sitios de glicosilación es una adaptación del virus a los hospedadores humanos. También cabe señalar que los autores no percibieron ningún cambio genético en la HA del virus de 1918 que explicaría su excepcional virulencia.
A diferencia de las cepas virulentas de la influenza aviar moderna, como los virus de influenza aviar A (H5) y (H7), la HA del virus de 1918 no poseía una mutación del «sitio de clivaje», que es un marcador genético reconocido de la virulencia, es decir la gravedad o nocividad de una enfermedad. La inserción de aminoácidos en el sitio de clivaje de la HA puede permitir que un virus de la influenza se reproduzca en tejidos fuera de sus células hospedadoras normales. Ante la ausencia de dichos marcadores, la Dra. Reid y sus socios investigadores concluyeron que probablemente hubo múltiples factores genéticos responsables de la gravedad del virus de 1918.

El Dr. Peter Palese, microbiólogo, y su equipo crearon los plásmidos utilizados por el Dr. Terrence Tumpey para reconstruir el virus de la pandemia de 1918. Palese tiene muchos logros, incluida la creación de los primeros mapas genéticos de los virus de influenza A, B y C, además de la definición del mecanismo utilizado por la mayoría de los actuales medicamentos antivirales contra la influenza. Foto por: Wikipedia (https://en.wikipedia.org/wiki/Peter_Palese)
Se publicó un documento de seguimiento en junio del 2000, titulado “Characterization of the 1918 “Spanish” Influenza Virus Neuraminidase Gene” que describió la secuenciación del gen de la neuraminidasa (NA) del virus de 1918.9 En un virus de la influenza, el gen de la neuraminidasa es responsable de la codificación de las proteínas superficiales de la NA del virus (ver la imagen anterior del virus para referencia). Las proteínas superficiales de la NA del virus de la influenza permiten que el virus de la influenza libere una célula infectada e infecte a las demás células. Por lo tanto, juegan un papel decisivo en la propagación del virus de la influenza. El autor notó que la NA también se ve afectada por el sistema inmunitario y que los anticuerpos contra la NA no previenen una infección, pero limitan considerablemente la capacidad de propagación del virus.
Cabe señalar que los autores pudieron realizar la secuenciación del código completo de la NA del virus de 1918 de la muestra del virus obtenida del cuerpo de “Lucy”. Así que nuevamente, el trabajo de Hultin resultó de gran utilidad. Los autores descubrieron que el gen de la NA del virus de 1918 compartía varias características estructurales y secuenciales con las cepas del virus de la influenza aviar y de mamíferos.9 El análisis filogenético sugirió que el gen de la NA del virus de 1918 fue localizado de manera intermitente entre mamíferos y aves, lo que sugiere que probablemente fue introducido entre los mamíferos inmediatamente antes de la pandemia de 1918. Además, la NA del virus de 1918 obtenida del cuerpo de Lucy sugirió que es bastante similar al predecesor de todas las muestras aisladas del virus de la influenza porcina y humana subsiguientes.9
En general, el análisis filogenético parecía indicar que la fuente principal de la NA del virus de 1918 era de naturaleza aviar, pero los autores no podían determinar el recorrido desde la fuente aviar hasta la forma pandémica final del virus. Respecto de las características genéticas de la NA que podría explicar la gravedad del virus de 1918, nuevamente los investigadores no fueron capaces de descubrir alguna característica de la NA de 1918 que haya contribuido a la virulencia del virus.9 Por ejemplo, en algunos de los virus de la influenza moderna, la pérdida del sitio de glicosilación en la NA en el aminoácido 146 (en WSN/33) contribuye a la virulencia y además da lugar a que el virus ataque el sistema nervioso de los ratones. Sin embargo, este cambio no se produjo en la NA del virus de 1918.
Después de este estudio, se publicó una serie de estudios adicionales con detalles de los hallazgos de cada uno de los genes remanentes del virus de 1918 (los virus de la influenza tienen 8 genes en total). En el 2001, un documento de Christopher Basler et al. publicado en Proceedings of the National Academic of Science (PNAS) describió la secuenciación del gen no estructural del virus de 1918.10 Un estudio del 2002 de Ann Reid et al. publicado en Journal of Virology describió la secuenciación del gen de la matriz del virus.11 Dos años más tarde, un estudio del 2004 publicado en Journal of Virology describió la secuenciación del gen nucleoproteico del virus de 1918.12 En 2005, los genes de la polimerasa del virus fueron secuenciados por Taubenberger et al. y descritos en un artículo de Nature.13 Este estudio final enmarcó un proceso de casi una década de secuenciación del genoma completo del virus de 1918.
Tras realizar la secuenciación de todo el genoma del virus de 1918, contaban con la información necesaria para reconstruir una versión viva del virus de 1918. Sin embargo, se necesitaba dar otro paso intermedio para iniciar el proceso de genética inversa, que consistía en crear plásmidos para cada uno de los ocho segmentos de genes del virus de 1918.
Esta tarea fue llevada a cabo por el Dr. Peter Palese, un reconocido microbiólogo, y el Dr. Adolfo Garcia-Sastre en Mount Sinai School of Medicine de la Ciudad de Nueva York. Un plásmido es una pequeña cadena circular de ADN que puede amplificarse (o reproducirse) en el laboratorio. Años atrás, el Dr. Palese ayudó a encauzar el uso de plásmidos en la genética inversa para generar un virus viable de la influenza. Las técnicas que desarrolló permitieron estudiar las relaciones entre la estructura y la función de los genes virales y estos esfuerzos allanaron el camino para utilizar las técnicas para reconstruir el virus de 1918. Cuando el Dr. Palese y sus colegas en Mount Sinai completaron la creación de plásmidos, los enviaron a los CDC para que se pudiera dar inicio al proceso oficial de reconstrucción.
La reconstrucción
La decisión de reconstruir el virus de la influenza pandémica más mortal del siglo XX fue tomada con especial cuidado y la atención puesta en la seguridad. Los funcionarios de gobierno sénior decidieron que la ubicación para la reconstrucción serían las oficinas centrales de los CDC en Atlanta. Los CDC realizaron dos escalones de aprobación: el primero por parte del Comité de Bioseguridad Institucional de los CDC y el segundo, del Comité de Cuidado Animal y Uso Institucional de los CDC, antes de que comience el trabajo de laboratorio. El trabajo se realizaría tomando estrictas precauciones de bioseguridad, incluidas las prácticas y los establecimientos mejorados de bioseguridad nivel 3 (BSL-3).

Una foto del Dr. Terrence Tumpey trabajando en condiciones de laboratorio mejoradas de BSL3. Esto incluye, entre otras cosas, el uso de una mascarilla de respiración con purificador de aire motorizado (PAPR, por sus siglas en inglés), guantes dobles, un traje y un gabinete de trabajo de bioseguridad clase II (BSC). Actualmente, el Dr. Tumpey es el jefe de la Subdivisión de Patogénesis e Inmunología de la División de Influenza de los CDC. Foto por: James Gathany – Biblioteca de imágenes de salud pública n.° 7989.
Existen cuatro niveles de bioseguridad que se corresponden con el grado de riesgo que representa la investigación, en el que 1 representa menos riesgo y 4 representa mayor riesgo. Cada nivel de bioseguridad también se corresponde con las prácticas y técnicas de laboratorio específicas, los requisitos de la capacitación del personal, el equipamiento de laboratorio y los establecimientos de laboratorios que son adecuados para que se lleven a cabo las operaciones. La exigencia de estas consideraciones, en la que 1 es la más baja y 4 es la más alta, está diseñada para proteger al personal que realiza el trabajo, al entorno y a la comunidad.
Cada nivel de bioseguridad incluye consideraciones para lo que se conoce como barreras «primarias» y «secundarias». Los ejemplos de barreras primarias incluyen gabinetes de seguridad, cámaras de aislamiento, guantes y batas, mientras que las barreras secundarias incluyen consideraciones tales como la construcción del establecimiento y los filtros HEPA para el tratamiento del aire en el laboratorio.
Un laboratorio BSL-3 con mejoras incluye una cierta cantidad de barreras primarias y secundarias, entre otras consideraciones. Por ejemplo, todo el personal debe usar mascarillas de respiración con purificador de aire motorizado (PAPR), guantes dobles, indumentaria quirúrgica, protectores de calzado y una bata quirúrgica. Además, deben darse una ducha antes de salir del laboratorio. Asimismo, todo el trabajo con los virus o los animales debe llevarse a cabo dentro de un gabinete de bioseguridad clase II (BSC) certificado y el flujo de aire que circula dentro del laboratorio es controlado y filtrado de modo que no puede escaparse del laboratorio.
Para la reconstrucción del virus de 1918, se crearon más reglas para regular los experimentos que se iban a realizar. Por ejemplo, para evitar confusiones y contaminación cruzada, el trabajo realizado en el virus de 1918 no podía llevarse a cabo junto con otros virus de la influenza.
Como parte de los aspectos de seguridad, la Oficina del Director de los CDC determinó que solo se le concedería permiso a una persona para ingresar al laboratorio y ser responsable de realizar la reconstrucción del virus de 1918. Esa persona fue el Dr. Terrence Tumpey, microbiólogo, quien contó con la aprobación de la Dra. Julie Gerberding, directora de los CDC, para llevar a cabo el proyecto. La reconstrucción del virus de 1918 fue aprobada por el Instituto Nacional de Alergias y Enfermedades Infecciosas de los Institutos Nacionales de la Salud (NIAID y NIH, por sus siglas en inglés, respectivamente), que financiaron parte del proyecto.
El Dr. Tumpey anteriormente fue microbiólogo del Departamento de Agricultura de los Estados Unidos (USDA) en el Southeast Poultry Research Laboratory en Athens, Georgia. Al principio de su carrera, solicitó una beca posdoctoral de la American Society of Microbiology (ASM) con la Dra. Jacqueline Katz, microbióloga y experta en influenza de los CDC, quien hace poco se retiró como Subdirectora de la División de Influenza de los CDC. Esta beca de dos años en la División de Influenza de los CDC marcaría el inicio de la carrera profesional del Dr. Tumpey en los CDC. Su empleo fue trasladado oficialmente a los CDC a fin de estudiar las consecuencias de los virus de la influenza en la salud humana, incluido el virus pandémico de 1918.
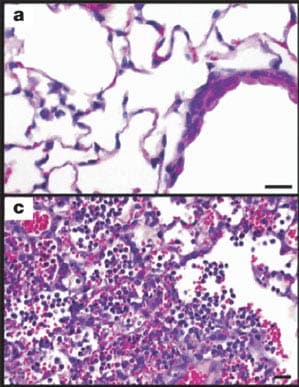
El virus de 1918 era extremadamente virulento. La imagen (a) muestra tejido pulmonar infectado por un virus de la influenza estacional humana H1N1. La imagen (c) muestra el impacto del virus de 1918 en el tejido pulmonar de un ratón. El virus de 1918 se reproduce rápidamente y causa enfermedades graves en los tejidos pulmonares de los ratones. En 1918, el virus también causó enfermedades graves en los pulmones de las personas infectadas. Foto por: CDC, Science.
El trabajo del Dr. Tumpey de reconstrucción del virus de 1918 comenzó durante el verano del 2005. Para evitar poner en riesgo a los colegas y al público en general, él tuvo que trabajar solo con el virus y fuera de horario, cuando sus colegas salían del laboratorio y se iban a sus casas. Era necesario pasar por el lector biométrico de huella digital para poder ingresar al laboratorio BSL-3E y solo se podía acceder a las cámaras frigoríficas donde almacenaban el virus a través del escáner de iris. El Dr. Tumpey debía tomar una dosis diaria de oseltamivir, un medicamento antiviral contra la influenza (a modo preventivo) como tratamiento profiláctico y como una precaución de seguridad adicional para evitar que contraiga una infección. En caso de que se infectara, se le informó que sería puesto en cuarentena y se le negaría entrar en contacto con el mundo exterior. Lo entendió y aceptó esta responsabilidad y sus consecuencias.
Mediante el uso de la genética inversa, el Dr. Tumpey tomó los plásmidos creados por el Dr. Palese para cada uno de los ocho segmentos de genes del virus de 1918 y los colocó en las células de riñones humanos. Luego los plásmidos les indicaron a las células que reconstruyan el ARN del virus completo de 1918. Durante varias semanas en julio del 2005, colegas y colaboradores le preguntaban al Dr. Tumpey si tenía el virus de 1918 y si ya había aparecido en el cultivo de células.
El día que el virus de 1918 apareció en su cultivo de células, el Dr. Tumpey supo que habían hecho historia al recuperar un virus histórico que se había extinguido. Más tarde ese mismo día, les envió a sus colegas y colaboradores un correo electrónico inspirado en una frase de Neil Armstrong con tono bromista, que simplemente decía: «Es un pequeño paso para el hombre, pero un gran salto para la humanidad». Entonces todos supieron que lo había logrado. El Dr. Tumpey se había convertido en el primer hombre que pudo reconstruir el virus completo de 1918. El próximo paso era estudiarlo y descifrar sus secretos letales.
Los estudios de laboratorio sobre el virus reconstruido de 1918 comenzaron a realizarse en agosto del 2005. Un informe de este trabajo, “Characterization of the Reconstructed 1918 Spanish Influenza Pandemic Virus”, se publicó en la edición del 7 de octubre del 2005 de Science.14 Para evaluar la patogenicidad del virus de 1918 (es decir, la capacidad del virus de causar una enfermedad en un organismo hospedador), se realizaron estudios en animales como ratones. Los ratones fueron infectados con el virus de 1918 y se tomaron medidas de morbilidad (es decir, descenso de peso, reproducción del virus y valores de dosis letal 50 %) que luego fueron documentadas. Para realizar una comparación, se infectaron a otros ratones con diferentes virus de la influenza que fueron diseñados a través de la genética inversa para tener diversas combinaciones de genes del virus de 1918 y los virus de influenza estacional A(H1N1) contemporáneos. Estos virus son llamados “virus recombinados”.
El virus completamente reconstruido de 1918 fue sorprendente en cuanto a su capacidad para reproducirse rápidamente, es decir, hacer copias de sí mismo y propagar la infección en los pulmones de ratones infectados. Por ejemplo, cuatro días después de la infección, la cantidad de virus de 1918 que se detectó en el tejido pulmonar de los ratones infectados fue 39 000 veces mayor que la que produjo uno de los virus de la influenza recombinados.14
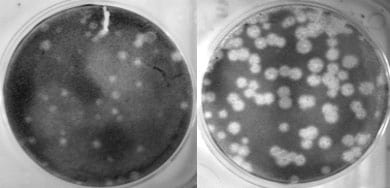
La imagen de la izquierda muestra la reproducción de un virus de la influenza estacional humana llamado Tx/91 en cultivo celular. La imagen de la derecha muestra cómo cuando el gen de la polimerasa (PB1) de este mismo virus es intercambiado por el del virus de 1918, la capacidad que tiene el virus de reproducirse (es decir, copiarse a sí mismo) es considerablemente mayor. Foto por: Terrence Tumpey, CDC.
Además, el virus de 1918 fue altamente letal en los ratones. Algunos ratones murieron al cabo de tres días a causa de la infección por el virus de 1918 y perdieron hasta el 13 % del peso corporal a los dos días de la infección por el virus de 1918. El virus de 1918 fue al menos 100 veces más letal que uno de los otros virus recombinados que fueron sometidos a prueba.14 Los experimentos demostraron que el gen de HA del virus de 1918 fue decisivo en cuanto a su gravedad. Cuando el gen de HA del virus de 1918 fue cambiado por el virus de influenza estacional humana A (H1N1) conocido como «A/Texas/36/91» o Tx/91 abreviado y combinado con los siete genes restantes del virus de 1918, el virus recombinado resultante no causó la muerte de los ratones infectados ni les provocó una pérdida de peso considerable.14
Se realizaron otros experimentos para determinar si la infección por el virus de 1918 podía propagarse a otros órganos vitales de los ratones, como el cerebro, el corazón, el hígado y el bazo. Las pruebas de laboratorio no detectaron el virus en estos órganos, lo que sugiere que el virus de 1918 no causaba una infección sistemática en sus víctimas.
Sin embargo, un efecto del virus de 1918 muy bien documentado fue un daño pulmonar rápido y severo. En 1918, a las víctimas del virus pandémico se les llenaron de líquido los pulmones y tuvieron un cuadro severo de neumonía e inflamación del tejido pulmonar. Dentro de los cuatro días posteriores a la infección, los ratones infectados por el virus de 1918 experimentaron complicaciones pulmonares similares, lo que sugiere que este era uno de los aspectos únicos de la gravedad del virus de 1918.14
También se estudió el impacto del virus de 1918 en el tejido pulmonar mediante el uso de una línea de células de pulmón humano (conocidas como células Calu-3). Se constató la cantidad de virus de influenza de 1918 que había en las células al cabo de 12, 16 y 24 horas posteriores a la infección y estos resultados se compararon con los producidos por los virus recombinados con una combinación de genes del virus de 1918 mezclados con genes de virus de la influenza estacional humana contemporáneos. Al igual que los experimentos con ratones, el virus de 1918 se multiplicó rápidamente y se propagó dentro de las células pulmonares humanas. Tanto es así que el virus de 1918 produjo 50 veces la cantidad de virus en células pulmonares humanas que uno de los virus comparados. Estos experimentos sugirieron que además de la HA, los genes de la polimerasa del virus de 1918 desempeñaron un papel decisivo en cuanto al nivel de contagio y virulencia del virus en tejido pulmonar humano.14

Un científico del laboratorio de los CDC «examina al trasluz» un huevo de gallina para ver el embrión en su interior. Foto por: James Gathany – Biblioteca de imágenes de salud pública n.° 10759.
Se realizó otra serie de experimentos para entender mejor los posibles orígenes aviares del virus de 1918. Los esfuerzos de secuenciación realizados anteriormente por los doctores Taubenberger y Reid habían sugerido que los segmentos de genes del virus de 1918 estaban más estrechamente relacionados con los virus de influenza aviar A(H1N1) que con los virus H1N1 encontrados en otros mamíferos. Los investigadores estaban interesados en saber si sería letal inyectar el virus de 1918 en huevos de gallina fertilizados, es decir, huevos de gallinas con embriones, como sucede con los virus de influenza aviar altamente patógenos de la actualidad.
Para encontrar la respuesta, se inocularon huevos de gallina fertilizados de 10 días con el virus de 1918. Se demostró que el virus de 1918 era letal para los embriones dentro de los huevos de gallina al igual que los efectos causados por los virus de influenza aviar H1N1 contemporáneos.14 Cabe señalar que experimentos comparativos en los que se utilizaron virus de influenza estacional humana A(H1N1) no mostraron este efecto nocivo en los embriones de pollos. Además, los virus de influenza recombinados que creó el Dr. Tumpey e incluían dos, cinco o siete genes del virus de 1918 no afectaron a los embriones de pollos.14 Al igual que los resultados de los estudios realizados en células pulmonares de humanos y ratones, estos experimentos con huevos de gallina fertilizados indicaron que los genes de la polimerasa y HA del virus de 1918 fueron decisivos en cuanto a su virulencia.
El trabajo realizado por el Dr. Tumpey y sus colegas de los CDC brindaron nueva información acerca de las propiedades que contribuyeron a la virulencia del virus de 1918. El Dr. Tumpey determinó que los genes virales de HA y PB1 fueron factores particularmente importantes en la gravedad y el riesgo de contagio del virus. No obstante, como demostraron sus experimentos que incluían virus de la influenza recombinados con algunos de, aunque no todos, los genes del virus de 1918, no fue un solo componente del virus de 1918 sino la combinación única de todos sus genes juntos lo que lo hizo tan peligroso.
Tumpey y sus colegas escribieron: “la constelación de los ocho genes juntos crearon un virus excepcionalmente virulento”.14 Ningún otro virus de la influenza humana sometido a prueba ha sido tan virulento. En ese sentido, el virus de 1918 era especial, un producto excepcionalmente mortal en cuanto a su naturaleza, evolución y capacidad de mezclarse entre las personas y los animales. Serviría como un presagio de la capacidad que tiene la naturaleza de producir futuras pandemias de diversos orígenes y de interés para la salud pública.
Lecciones del pasado

Las condiciones de hacinamiento y el traslado de las tropas durante la Primera Guerra Mundial probablemente contribuyeron en la propagación del virus de 1918 alrededor del mundo. (Foto por: www.museumsyndicate.com/item.php?item=56784#)
Desde 1918, el mundo ha experimentado otras tres pandemias en 1957, en 1968 y más recientemente en el 2009. Estas pandemias subsiguientes fueron menos severas y causaron índices de mortalidad mucho más bajos que los de la pandemia de 1918.2,3,4 La pandemia de influenza H2N2 1957 y la pandemia H3N2 1968 dejaron un saldo estimado de 1 millón de muertes a nivel mundial, mientras que la pandemia de influenza H1N1 2009 provocó un poco menos de 0.3 millón de muertes durante su primer año.3,4 Esto quizás plantea la pregunta sobre si en los tiempos modernos podría ocurrir una pandemia de gravedad similar a la de 1918.
Muchos expertos creen que sí. Un virus en particular ha generado una gran preocupación y ha acaparado la atención a nivel internacional: el virus de la influenza aviar A(H7N9) de China. El virus de la influenza H7N9 ha causado hasta ahora 1 568 infecciones en humanos en China con un índice de mortalidad del 39 % desde el 2013. Sin embargo, no ha tenido la capacidad de propagarse rápidamente entre las personas. De ser así, los expertos consideran que hubiera causado una pandemia con un nivel de gravedad similar al de la pandemia de 1918. Hasta ahora, solo ha demostrado una capacidad limitada para propagarse entre las personas. La mayoría de las infecciones en humanos por este virus se debieron a una exposición a las aves.
No obstante, al considerar la posibilidad de una pandemia de gravedad muy alta en la era moderna, es importante reflexionar sobre los avances médicos, científicos y sociales que han acontecido desde 1918 y reconocer que existen varias maneras de mejorar aún más la preparación a nivel mundial para enfrentar la próxima pandemia.
Más allá de las propiedades del virus en sí mismas, muchos otros factores contribuyeron con la virulencia de la pandemia de 1918. En 1918, el mundo aún estaba involucrado en la Primera Guerra Mundial. El traslado y la movilización de las tropas hicieron que una gran cantidad de personas estuviera en contacto directo y conviviera hacinada. Los servicios de salud escaseaban y hasta el 30 % de los médicos fue enviado al servicio militar.3
Además, la tecnología de los servicios médicos y las contramedidas de aquel momento eran limitadas o prácticamente no existían. En ese entonces no existían pruebas de diagnóstico que detectaran una infección por el virus de la influenza. De hecho, los médicos no sabían que existían virus de la influenza. Muchos expertos de la salud de aquel entonces pensaban que la pandemia de 1918 era causada por una bacteria llamada «bacilo de Pfeiffer» que actualmente se la conoce como «Haemophilus influenzae».
Por aquel entonces, tampoco existían vacunas contra la influenza e incluso aún no se habían fabricado los antibióticos. Por ejemplo, la penicilina no se descubrió hasta 1928. Asimismo, tampoco había medicamentos antivirales contra la influenza. Las medidas de cuidados críticos, como los cuidados intensivos y la asistencia respiratoria mecánica, no existían en 1918.4 Sin contramedidas médicas ni tratamientos, los médicos tenían muy pocas opciones de tratamiento además de los cuidados paliativos.3
En cuanto a la planificación ante la pandemia a nivel local, estatal y nacional, en 1918 no existían planes coordinados contra la pandemia. Algunas ciudades se las arreglaron para implementar medidas de mitigación en la comunidad, como el cierre de escuelas, la prohibición de reuniones públicas y la emisión de órdenes de cuarentena o aislamiento, pero el gobierno federal no tenía una función centralizada para ayudar a planificar o iniciar estas intervenciones durante la pandemia de 1918.3
Actualmente, se han hecho importantes avances en las áreas de tecnología de la salud, vigilancia de la enfermedad, atención médica, medicamentos, vacunas y planificación para la pandemia. Ahora se fabrican vacunas contra la influenza y se actualizan anualmente, y además se recomienda la vacunación anual contra la influenza a todas las personas a partir de los 6 meses de edad en adelante. Los medicamentos antivirales que existen en la actualidad sirven para tratar los casos de influenza y, en caso de exposición al virus, también pueden usarse para la profilaxis (prevención). Cabe destacar que muchos de los diferentes antibióticos que actualmente están disponibles pueden utilizarse para tratar infecciones bacterianas secundarias.
Ahora hay pruebas de diagnóstico disponibles para detectar la influenza, las cuales siguen mejorando con el paso del tiempo. Las pruebas de detección rápida actuales, como las RIDT, ofrecen resultados al cabo de 15 minutos y tienen una sensibilidad del 50-70 %. Recientemente, se pusieron a disposición nuevas «pruebas moleculares de detección rápida» que son mucho más precisas y oportunas que las RIDT. Tan importante como estos avances en las pruebas de diagnóstico son las mejoras que se realizaron en la capacidad de los laboratorios para realizar pruebas tanto dentro de los Estados Unidos como en resto del mundo.
El Sistema de respuesta y vigilancia de la influenza a nivel mundial (GISRS) de la Organización Mundial de la Salud (OMS) es una red mundial de vigilancia de la influenza que monitorea los cambios en los virus de la influenza estacional y además monitorea la aparición de nuevos virus de la influenza (es decir, nuevos en seres humanos), muchos de los cuales se originan en las poblaciones de animales. A través de las interacciones entre los seres humanos y los animales y las exposiciones ambientales, estos virus pueden causar infecciones en humanos. Los CDC en Atlanta son uno de los seis Centros de Colaboración de Referencia e Investigación de la Influenza de la OMS (junto a otros en Australia, China, Japón y el Reino Unido). Los Centros de Colaboración de la OMS toman muestras de virus de la influenza obtenidas de especímenes respiratorios de pacientes de todo el mundo y cuentan con el apoyo de 143 Centros Nacionales para la Influenza en 114 países miembros de la OMS.3
Expandir la capacidad de vigilancia de la influenza y las pruebas de laboratorio en todo el mundo ha sido un importante enfoque de los esfuerzos de preparación ante una pandemia. En el 2004, los CDC iniciaron una iniciativa para el desarrollo de capacidades de vigilancia a nivel internacional que incluyó un respaldo financiero por un lapso de 5 años con el propósito de mejorar las pruebas de diagnóstico de laboratorio y la vigilancia de la enfermedad similar a la influenza (ILI, por sus siglas en inglés) y la infección respiratoria aguda (IRA) en 39 países participantes.
En el 2008, los CDC establecieron los Recursos de reactivos internacionales (IRR, por sus siglas en inglés), que proporcionan reactivos a los laboratorios de todo el mundo para identificar los virus de influenza estacional A y B además de los nuevos virus de influenza A. Durante la pandemia de influenza H1N1 2009, los IRR distribuyeron una nueva prueba de reacción en cadena de la polimerasa (PCR, por sus siglas en inglés) para detectar el virus de influenza H1N1 2009 creada por los CDC para los laboratorios de salud pública nacionales y los laboratorios internacionales en menos de 2 semanas después de que se identificara el primer virus de influenza H1N1 2009. Esto permitió mejorar considerablemente la capacidad de la comunidad mundial de vigilancia de la influenza para hacer un seguimiento de la propagación del virus.3
Como parte de las Regulaciones internacionales de salud (IHR, por sus siglas en inglés) de la OMS, los países deben notificar a la OMS dentro de las 24 horas de cada caso de infección en seres humanos causado por un nuevo subtipo del virus de influenza A. Este requisito está diseñado para ayudar a identificar rápidamente los virus emergentes con potencial pandémico.
Desde el 2010, los CDC han utilizado su Herramienta de evaluación del riesgo de influenza (IRAT, por sus siglas en inglés) para evaluar y calificar los nuevos virus de influenza A emergentes y otros virus de posible interés para la salud pública. La calificación otorgada por la IRAT responde a dos preguntas: 1) ¿Cuál es el riesgo de que un virus que es nuevo entre los seres humanos pueda causar un contagio sostenido entre humanos? y 2) ¿Cuál es la posibilidad de que el virus impacte considerablemente en la salud pública en caso de que adquiera la capacidad de propagarse de manera efectiva de persona a persona? Los resultados de la IRAT han permitido que los expertos de salud pública dirijan los recursos de preparación para una pandemia ante las principales amenazas de enfermedades y prioricen la selección de los virus candidatos para la vacuna y el desarrollo de vacunas prepandémicas contra los virus emergentes con el potencial de causar una pandemia severa.
Cuando se fabrican vacunas prepandémicas, son almacenadas en la Reserva Nacional Estratégica, junto con las mascarillas, los medicamentos antivirales y otros materiales que pueden utilizarse en caso de una pandemia.
Todos estos recursos, herramientas, tecnologías, programas y actividades son excelentes herramientas para la planificación ante una pandemia; de hecho, la planificación para una pandemia ha mejorado considerablemente desde 1918. En los Estados Unidos, el Departamento de Salud y Servicios Humanos (HHS) tiene un plan nacional contra la influenza pandémica y este plan fue actualizado en el 2017 La Organización Mundial de la Salud (OMS) ha publicado instrucciones para que los países puedan crear sus propios planes nacionales para una pandemia, además de una lista de verificación para la administración de riesgos e impacto de la influenza pandémica.3
Los planificadores también tenían acceso a otros materiales. Por ejemplo, en el 2014, los CDC publicaron un marco para la pandemia con seis intervalos dentro de la curva pandémica. Cada intervalo ayuda a priorizar la recolección de datos, los recursos y las intervenciones gubernamentales, además de otras actividades importantes durante la pandemia. Además, los expertos de los CDC han creado un marco de evaluación de la gravedad de una pandemia que utiliza datos para asignarles calificaciones de gravedad y transmisibilidad a las pandemias. La herramienta es útil para los fines de planificación y para determinar las mitigaciones correspondientes según la gravedad de una pandemia. Además, las directrices para las intervenciones no farmacéuticas, como el cierre de escuelas y la prohibición de reuniones sociales masivas, han sido establecidas y revisadas para ser utilizadas durante una pandemia.
Si bien todos estos planes, recursos, productos y mejoras reflejan ese avance significativo que se ha logrado desde 1918, aún quedan brechas por cerrar y una pandemia grave podría devastar a poblaciones de todo el mundo. En 1918, la población mundial era de 1.8 mil millones de personas. Cien años después, aumentó la población mundial a 7.6 mil millones de personas en el 2018.3 Del mismo modo que aumentó la población humana, también aumentó la cantidad de cerdos y aves para poder alimentar a los humanos. Al haber una mayor cantidad de hospedadores aumentan las posibilidades de que nuevos virus de la influenza provenientes de cerdos y aves se propaguen, evolucionen e infecten a seres humanos. El traslado de personas y mercancías a nivel mundial también aumentó, por lo que la amenaza más reciente de una enfermedad está a un vuelo internacional de distancia. Debido a la movilidad y la expansión de la población de seres humanos, incluso patógenos exóticos, como el virus del Ébola, que anteriormente afectaban solo a las personas que vivían en la selva africana, ahora han encontrado la manera de introducirse en áreas urbanas causando brotes de mayor magnitud.
Si actualmente ocurriera una pandemia severa, como la que ocurrió en 1918, probablemente volvería a hacer colapsar la infraestructura de cuidado de salud, tanto en los Estados Unidos como en el resto del mundo. Los hospitales y consultorios médicos tendrían que esforzarse para cubrir la demanda por la cantidad de pacientes que requerirían de atención médica. Algo así requeriría de un incremento importante en la fabricación, la distribución y el suministro de medicamentos, productos y equipos médicos como respiradores mecánicos. Se complicaría el funcionamiento de escuelas y empresas e incluso podrían verse afectados servicios básicos como la recolección de residuos.
La mejor defensa contra la influenza sigue siendo la vacuna contra la influenza, pero incluso en la actualidad las vacunas contra la influenza enfrentan varios retos. Uno de los retos es que las vacunas contra la influenza suelen tener una efectividad moderada, incluso cuando existe una buena correspondencia con los virus en circulación. Pero quizás el mayor reto es el tiempo que lleva crear una nueva vacuna contra la amenaza emergente de una pandemia. Por lo general, la selección y la fabricación de una nueva vacuna demora alrededor de 20 semanas.
Durante la pandemia de influenza H1N1 2009, las primeras dosis de la vacuna contra la pandemia tardaron hasta 26 semanas en estar disponibles después de que se tomara la decisión de fabricar una vacuna monovalente.3 Como resultado, la mayoría de las vacunaciones en los Estados Unidos se realizaron después de que la actividad de la influenza H1N1 2009 alcanzara su punto máximo. El Plan contra la influenza pandémica del HHS tiene el objetivo de reducir el plazo de tiempo para poder fabricar una vacuna contra la influenza pandémica entre 20 y 12 semanas, pero lograrlo es todo un reto.
Una solución posible es fabricar una vacuna de amplia protección con efectos más duraderos. La creación de una «vacuna universal» escapa al alcance de los destacados científicos del mundo, pero a futuro podría hacerse realidad. Mientras tanto, los funcionarios de la salud buscan sacar el máximo provecho de las actuales y nuevas tecnologías para la vacuna contra la influenza, como las vacunas recombinadas y a base de células, las cuales no dependen del suministro de huevos de gallina como las vacunas tradicionales y tienen el potencial de ser fabricadas con mayor rapidez.
Otro problema con la vacuna es la capacidad mundial inadecuada para fabricar vacunas contra la influenza en forma masiva. Se estimó que la capacidad mundial de la vacuna contra la influenza pandémica era de 6.4 mil millones de dosis en el 2015, pero esto no es suficiente ni para cubrir a la mitad de la población mundial si se requieren de dos dosis de la vacuna para protegerse contra la pandemia.3
Otros retos a nivel mundial incluyen la capacidad de vigilancia, la infraestructura y la planificación para una pandemia. La mayoría de los países que reportan a la OMS aún carecen de un plan nacional para una pandemia y la capacidad de los cuidados clínicos e intensivos, especialmente en los países de bajos recursos, sigue siendo inadecuada como para satisfacer la demanda de una pandemia severa.3 En el 2005, se crearon indicadores en las Regulaciones internacionales de salud (IHR) para que los países mejoren su capacidad de respuesta ante las emergencias de salud pública, pero en el 2016 solo un tercio de los países cumplía con los requisitos.3
Todos estos problemas demuestran la necesidad de hacer algo más, tanto en los Estados Unidos como en el resto del mundo, para estar preparados para la próxima pandemia. El 7 de mayo del 2018, The Rollins School of Public Health en Emory University, conjuntamente con los Centros para el Control y la Prevención de Enfermedades, organizó un simposio de un día con motivo de la conmemoración del 100.° aniversario de la pandemia de influenza de 1918. El evento contó con la presencia de expertos académicos y del gobierno que debatieron sobre las amenazas actuales de una pandemia y el futuro de la preparación ante una pandemia y la prevención y el control de la influenza. Los expertos en la influenza de los Estados Unidos y del mundo que asistieron a la reunión estuvieron de acuerdo en que aún enfrentamos varios retos para prepararnos para una futura pandemia de influenza, pero parte de la solución consiste en reconocer estos retos y trabajar en forma conjunta con el resto del mundo para enfrentarlos.
Referencias
- P. Johnson and J. Mueller. Updating the accounts: global mortality of the 1918-1920 «Spanish» influenza pandemic. Bull. Hist. Med. 2002. 76(1): 105-115. ícono de sitio externo
- Barbara Jester et al. Historical and clinical aspects of the 1918 H1N1 pandemic in the United States. Virology 2019. 527: 32-37.ícono de sitio externo
- Barbara Jester et al. Readiness for Responding to a Severe Pandemic 100 Years After 1918. Am J Epidemiol. July 2018. 187(12): 2596-2602. DOI: 10.11/aje/kwy165.ícono de sitio externo
- Barbara Jester et al. 100 Years of Medical Countermeasures and Pandemic Influenza Preparedness. Am J Public Health. 2018; 108: 1469-1472. Doi:10.21/AJPH.2018.304586.ícono de sitio externo
- NYT Magazine: Why revive a deadly flu virus? https://www.nytimes.com/2006/01/29/magazine/why-revive-a-deadly-flu-virus.htmlícono de sitio externo.
- K. Taubenberger, Ann Reid, Amy Krafft et al. Initial Genetic Characterization of the 1998 “Spanish’ Influenza Virus. Science. Mar 1997; Vol 275, Issue 5307, pp 1793-1796ícono de sitio externo.
- San Francisco Chronicle: The Virus detective / Dr. John Hultin has found evidence of the 1918 flu epidemic that had eluded experts for decades https://www.sfgate.com/magazine/article/The-Virus-detective-Dr-John-Hultin-has-found-2872017.phpícono de sitio externo
- Ann Reid et al. Origin and evolution of the 1918 «Spanish» influenza virus hemagglutinin gene PNAS. February 1999. Vol 96. Pp. 1651-1656.ícono de sitio externo
- Ann Reid et al. Characterization of the 1918 «Spanish Influenza Virus Neuraminidase Gene. PNAS. Vol. 97. No. 12. June 2000. Pp. 6785-6790ícono de sitio externo.
- Christopher Basler et al. «Sequence of the 1918 pandemic influenza virus nonstructural gene (NS) segment and characterization of recombinant viruses bearing the 1918 NS genes» PNAS. Vol. 98. No. 5. February 2001 Pp. 1746-2751.ícono de sitio externo
- Ann Reid et al. «Characterization of the 1918 «Spanish» Influenza Virus Matrix Gene Segment» Journal of Virology. Nov 2002 Vol. 76. No. 21. pp. 10717-10723ícono de sitio externo.
- Ann Reid et al. «Novel Origin of the 1918 Pandemic Influenza Virus Nucleoprotein Gene» Journal of Virology. Nov 2004. Vol. 78, No. 22. Pp. 12462-12470ícono de pdfícono de sitio externo.
- Jeffery K. Taubenberger et al. «Characterization of the 1918 influenza virus polymerase genes» Nature. October 2005. Vol 437 (6). Pp. 889-893ícono de sitio externo.
- Tumpey TM, Baster CF, Aguilar PV, et al. Characterization of the Reconstructed 1918 Spanish Influenza Pandemic Virus” Science. October 2005. 310(5745): 77-80.ícono de sitio externo
Recursos relacionados
- Terrence Tumpey et al. «Existing antivirals are effective against influenza viruses with genes from the 1918 pandemic virus.» PNAS. October 2002. Vol. 99. No. 21 pp. 13849-13854ícono de sitio externo.
- Terrence Tumpey et al. «Pathogenicity and immunogenicity of influenza viruses with genes from the 1918 pandemic virus. PNAS. March 2004. Vol. 101 No. 9. Pp. 3166-3171.ícono de sitio externo
- Gibbs MJ, Armstrong JS, Gibbs AJ, et al. «Recombination in the Hemagglutinin Gene of the 1918 «Spanish Flu» Science. September 2001. Vol 293 (5536). Pp. 1842-1845ícono de sitio externo.
- Laurel Glaser et al. «A Single Amino Acid Substitution in 1918 Influenza Virus Hemagglutinin Changes Receptor Binding Specificity» Journal of Virology. Sept 2005. Vol 79. No. 17. Pp. 11533-11536ícono de sitio externo.
- Ervin Fodor et al. «Rescue of Influenza A Virus from Recombinant DNA» Journal of Virology. Nov 1999. Vol 73. No. 11. Pp. 9679-9682ícono de sitio externo.
- Darwyn Kobasa et al. «Enhanced virulence of influenza A viruses with the haemagglutinin of the 1918 pandemic virus.» Nature. October 2004. Vol 431 pp. 703-707ícono de sitio externo.
- Masato Hatta et al. «Molecular Basis for High Virulence of Hong Kong H5N1 Influenza A Viruses» Science. September 2001. Vol 293. Pp. 1840-1842ícono de sitio externo.
- Peter Palese Bio – Icahn School of Medicine at Mount Sinai: http://labs.icahn.mssm.edu/paleselab/ícono de sitio externo
Descargo de responsabilidad: Es posible que en este sitio encuentre algunos enlaces que le lleven a contenido disponible sólo en inglés. Además, el contenido que se ha traducido del inglés se actualiza a menudo, lo cual puede causar la aparición temporal de algunas partes en ese idioma hasta que se termine de traducir (generalmente en 24 horas). Llame al 1-800-CDC-INFO si tiene preguntas sobre la influenza estacional, cuyas respuestas no ha encontrado en este sitio. Agradecemos su paciencia.
Esta página fue revisada el 17 de diciembre del 2019